La replicazione del DNA: il segreto della vita
L’importanza della replicazione
La replicazione del DNA è uno degli eventi principali del ciclo cellulare e la cosa che più sorprende, è che avviene sempre con la stessa meticolosa precisione. I sistemi di replicazione sono così accurati che il margine di errore stimato per questo processo, è di circa un nucleotide ogni 10^9 nuovi aggiunti. Se si volesse fare un paragone il ruolo svolto dalla replicazione del DNA, è simile alla ristesura di un testo: essa ha il compito di assicurare la riproduzione dell’informazione genetica; in altre parole permette al nostro corpo, di parlare sempre lo stesso linguaggio e di essere letto sempre allo stesso modo.Senza gli eventi di replicazione del DNA nessun gene avrebbe modo di esprimersi con il linguaggio delle basi azotate e di conseguenza nessuna informazione, riguardante la materia di cui siamo composti, potrebbe essere espressa e tramandata. In assenza degli eventi di replicazione, dunque, non ci sarebbe vita.
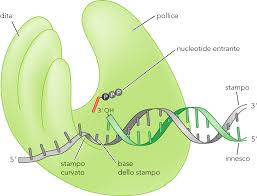
Figura 1 – struttura a “mano” della DNA-polimerasi [Fonte La replicazione del DNA]
La DNA-polimerasi
Questo enzima è sicuramente l’attore principale della replicazione del DNA. Il compito a cui assolve la DNA-polimerasi, è appunto “polimerizzare” cioè aggiungere nuovi nucleotidi al filamento nascente di DNA. I nucleotidi aggiunti, sono dNTPs (deossinucleotidi trifosfato) contenenti tre gruppi fosfato. Ogni qual volta un nucleotide è incorporato dalla polimerasi, dei tre gruppi fosfato che lo compongono due sono liberati per poi essere trasformati in pirofosfato da una pirofosfatasi e uno invece, resta attaccato alla base azotata. Il fatto che venga liberato pirofosfato, rende la replicazione del DNA un processo accoppiato perché coinvolge un’altra reazione chimica che la porta ad essere termodinamicamente favorita.
La DNA-polimerasi, ha una struttura che può essere paragonata ad una mano semichiusa e per questa ragione, sono stati individuati tre domini che hanno acquisito dei nomi singolari:

Figura 2 – Tabella riassuntiva di tutte le DNA – polimerasi operanti negli organismi [Fonte SlidePlayer]
Palmo: è formato da b-foglietti e contiene gli elementi che compongono il sito catalitico dell’enzima. Il compito di questo dominio è verificare il corretto appaiamento tra basi azotate e lo fa interagendo con il solco minore del DNA, solo in caso di appaiamenti corretti.
Dita: è un dominio formato da residui amminoacidici di Arginina, Lisina e Tirosina. All’entrata del dNTP le dita ne favoriscono la polimerizzazione, dal momento che piegano il filamento stampo a 90° in modo da avvicinare il nucleotide da aggiungere.
Pollice: ha il compito di mantenere nella giusta posizione la polimerasi interagendo con il DNA neo-sintetizzato.
Per poter procedere alla replicazione del DNA la polimerasi, ha bisogno di un innesco che funga da punto di partenza per l’enzima. L’innesco è chiamato primer, esso è sintetizzato dall’enzima “primasi” e nel processo di replicazione in vivo, è un breve filamento a RNA. L’unione del primer con il filamento da sintetizzare è definita “giunzione innesco-stampo” o “primer template”.
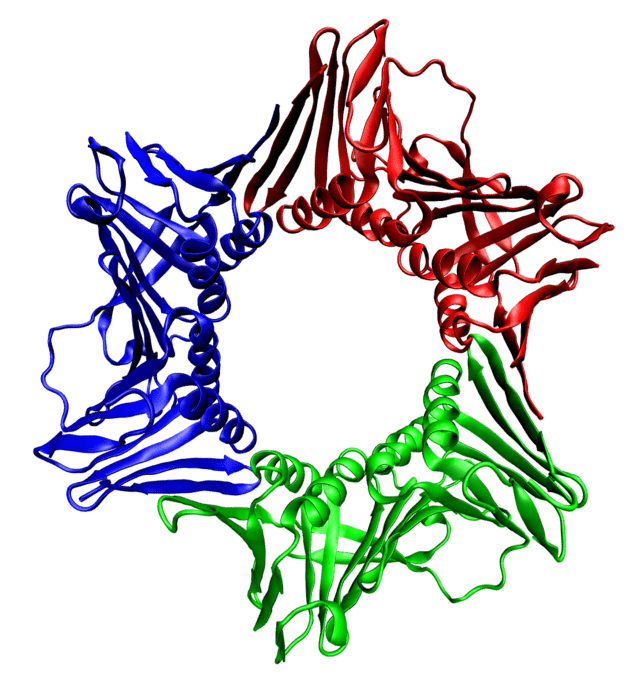
Figura 3 – Struttura ad anello delle Sliding Clamps [Fonte Wikiwand]
La classificazione delle DNA-polimerasi
In realtà della DNA-polimerasi non si può parlare al singolare perché in base al fatto che la replicazione del DNA avvenga in eucarioti o procarioti, intervengono enzimi appartenenti a famiglie diverse. Nella tabella seguente sono riassunte le principali classi di polimerasi:
Dalla tabella riportata si evince che alcune DNA-polimerasi, sono dotate di “proofreading activity”. Questo meccanismo solitamente avviene in direzione 3’-5’ ossia in senso contrario al verso di polimerizzazione dell’enzima. Quando la polimerasi riconosce un missmatch (corretto appaiamento mancato) diminuisce la sua processività legando con meno affinità il primer template. Questo favorisce l’escissione del nucleotide sbagliato e l’inserimento della base corretta.
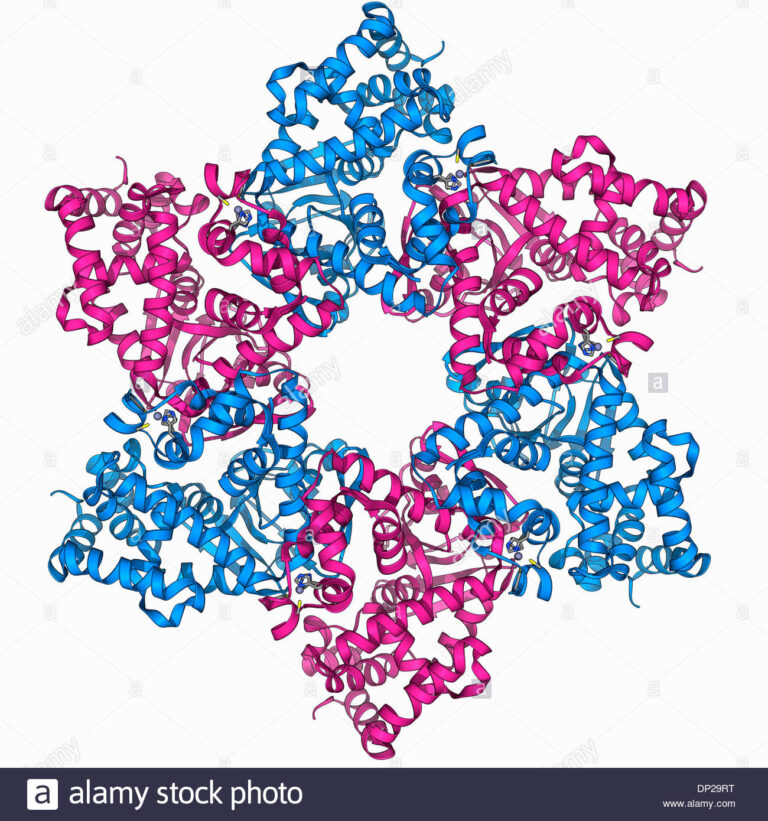
Figura 4 – struttura molecolare dell’elicasi [Fonte Alamy]
In generale di tutti gli enzimi riportati nello schema precedente quelli che partecipano maggiormente alla replicazione del DNA sono le “polimerasi a, d, e” (non processive) per quanto riguarda gli eucarioti, e la polimerasi III (altamente processiva) nei procarioti.
Di norma la DNA-polimerasi tenderebbe a staccarsi dal filamento e per questo motivo intervengono le sliding clamps (presenti sia in eucarioti che procarioti), proteine con il compito di assicurare l’enzima al DNA. Queste molecole proteiche, dalla forma ad anello, sono posizionate da un elemento chiamato “caricatore delle sliding clamp” e scorrono lungo l’acido nucleico grazie alla presenza di acqua. Dal momento che fissano meglio la polimerasi allo stampo, le sliding clamps aumentano di molto la processività della replicazione.
I sistemi di controllo della DNA-polimerasi
Per catalizzare il corretto appaiamento la DNA-polimerasi si avvale principalmente di tre vie di controllo:
Posizione in entrata dei nucleotidi: l’enzima discrimina la geometria dell’appaiamento e non la correttezza del nucleotide in entrata. Soltanto quando avviene un corretto appaiamento le basi si trovano spazialmente e geometricamente in posizione esatta, per poter entrare in modo preciso nel sito catalitico dell’enzima. Quanto detto è un esempio di selezione cinetica, presente in enzimi che aumentano di molto la velocità del processo solo in presenza del corretto substrato.
Differenziamento tra dNTP e rNTP: un altro compito dell’enzima è quello di differenziare i nucleotidi appartenenti al DNA da quelli che sono usati negli appaiamenti dell’RNA. Questo tipo di controllo fa fede ad un’esclusione sterica: solo se nel sito catalitico entra un dNTP il 3’OH e il fosfato del nucleotide si trovano vicini e permettono la polimerizzazione.
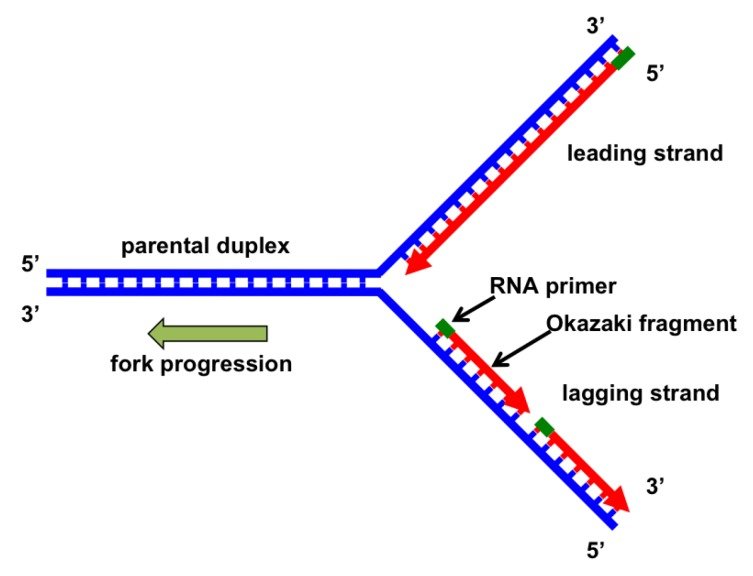
Figura 5 – rappresentazione della forca replicativa [Fonte mdpi]
La forca replicativa
Affinché la replicazione del DNA possa avvenire l’acido nucleico deve essere a singolo filamento per permettere l’interazione ai complessi di replicazione. L’enzima che si occupa della separazione dei filamenti è l’elicasi.
Le elicasi sono proteine esameriche che sfruttando l’energia prodotta dall’idrolisi dell’ATP, separano i due filamenti di DNA formando due ssDNA (DNA a singolo filamento). Ciascuna delle sei subunità che li compone lega lo scheletro fosfato del DNA e due molecole di deossiribosio, permettendo all’acido nucleico di interagire con il sito catalitico dell’enzima.
Nello svolgere il loro compito, le elicasi sono coadiuvate da un’altra classe enzimatica: le girasi.
Queste ultime appartengono alla classe delle topoisomerasi II e rimuovono i super avvolgimenti caratteristici della configurazione del DNA; così facendo stabilizzano l’acido nucleico in modo da rendere più efficace la separazione dei filamenti.
Il punto in cui le sequenze nucleotidiche complementari iniziano a dividersi è detto “forca replicativa” dal momento che ha come risultato, la formazione di due filamenti antiparalleli che fungono da stampo per i nuovi da sintetizzare.
Basandosi sul senso di lettura dei due filamenti, si possono denominare in modo specifico come:
LEADING STRAND, detto anche “filamento continuo” o “filamento veloce”. Ha direzione 3’-5’ e prevede un primer con orientamento 5’-3’ che permette la polimerizzazione immediata da parte della DNA-polimerasi.
LAGGING STRAND, detto anche “filamento discontinuo” o “filamento lento”. Procede in senso 5’-3’ e prevede l’azione di primer con orientamento 3’-5’. In questo caso la DNA-polimerasi deve attendere che ci sia sufficiente stampo per avviare la replicazione, si serve perciò di più primer.
Come si evince dalla figura riportata, nel filamento discontinuo ogni primer usato dalla polimerasi è collocato all’inizio di una sequenza a singolo filamento, successiva ad un tratto appena sintetizzato e perciò a doppio filamento. Si creano dei piccoli frammenti di DNA a doppio filamento, non continui tra loro e considerati degli intermedi di replicazione che prendono il nome di “frammenti di Okazaki”. Questi tratti discontinui di DNA, verranno infine uniti grazie all’azione di una ligasi.
I filamenti separati, tenderebbero a riunirsi velocemente per l’insieme di forze che li rende complementari. A questo proposito intervengono le proteine SSB con il compito di mantenere aperta la forca replicativa. Queste proteine agiscono con un meccanismo di legame cooperativo: la presenza una SSB ne richiama altre.

Figura 6 – struttura dell’oloenzima procaroitico [Fonte DocPlayer]
Lo switch delle polimerasi e l’Oloenzima in E. coli
La replicazione del DNA ha bisogno di essere svolta in modo quanto più processivo possibile e per questo motivo, in realtà, in questo meccanismo intervengono contemporaneamente più polimerasi. Nel caso degli eucarioti la prima DNA-polimerasi che interviene è la “polimerasi a”, costituita da quattro subunità di cui due hanno attività polimerasica e le restanti due hanno attività primasica.
La polimerasi di tipo “a” si associa subito ed inizia la polimerizzazione delle nuove catene, subito dopo è però rimpiazzata dalle “polimerasi d” ed “e” che sono più rapide e hanno la capacità di lavorare contemporaneamente in quanto la “polimerasi d” si occupa di copiare il filamento discontinuo mentre la “polimerasi e” sintetizza il filamento continuo.
Il fenomeno dello scambio tra questi enzimi è lo “switch delle polimerasi”.
Nel caso dei procarioti interviene invece un particolare complesso definito “oloenzima” e identificato per la prima volta in E. coli. Esso è formato da due DNA-polimerasi III, legate ad un caricatore delle sliding clamps mediante dei linker di proteina T. In questo modo le due polimerasi possono lavorare contemporaneamente su entrambi i filamenti e sono assistite nella replicazione, dalle sliding clamp che ne aumentano la processività.
Il modello del replicone
Gli scienziati analizzando la replicazione del DNA hanno messo a punto il “modello del replicone“. Questo sistema di studio si compone di:
Replicatore: è l’insieme di sequenze nucleotidiche che dirigono l’inizio della replicazione. Queste sequenze sono caratterizzate dall’abbondanza in accoppiamenti adenina-timina e da un sito di legame per i complessi di replicazione.
Iniziatore: è una proteina sequenza-specifica che riconosce il replicatore e attiva il processo di replicazione.
Nei procarioti il replicatore è la sequenza ORI-C ed è riconosciuta dall’iniziatore DNA a.
Negli eucarioti il replicatore è costituito da due sequenze che sono l’elemento A e l’elemento B1, entrambe riconosciute dall’iniziatore ORC.
L’inizio della replicazione del DNA
L’inizio della replicazione del DNA nei procarioti prevede il legame dell’iniziatore DNA a alla sequenza di inizio ORI-C. Il legame tra questi due elementi richiama un caricatore delle elicasi che assembla l’elicasi attorno alla doppia elica di DNA grazie all’ idrolisi di ATP. Questo evento porta all’intervento della primasi che si occupa di sintetizzare il primer a RNA; la formazione dell’innesco favorisce il distacco del caricatore delle elicasi in modo che l’elicasi possa iniziare a svolgere la doppia elica.
Non appena il DNA inizia ad essere scisso, la polimerasi III oloenzima si associa alla giunzione innesco-stampo iniziando così la replicazione. La forca replicativa si estende fin quando non incontra la fine del filamento stampo.
Negli eucarioti il processo si svolge essenzialmente allo stesso modo ma con dei sistemi di controllo più stringenti, perché il singolo cromosoma venga replicato una ed una sola volta. Questo problema si pone perché un cromosoma può possedere più origini di replicazione potenzialmente riconoscibili dai complessi replicativi. La cellula inizia a prepararsi per affrontare la replicazione del DNA in fase G1 del ciclo cellulare ma solo in fase S avviene la sintesi di nuovo acido nucleico.
Così come avviene nei procarioti, anche negli eucarioti l’iniziatore si lega al replicatore richiamando l’intervento del caricatore delle elicasi. In realtà in questo caso, le elicasi sono assemblate in fase G1 e restano ferme fino all’inizio della fase S.
A bloccare l’azione di questi enzimi prima del tempo sono le CDK e DDK, due famiglie di proteine chinasi-dipendenti che solo se presenti ad alte concentrazioni (come avviene esclusivamente in fase S del ciclo cellulare) favoriscono l’attivazione delle elicasi.
Da questo momento in poi la replicazione procede allo stesso modo di quanto descritto per i procarioti con l’accortezza di ricordare che al posto della DNA-polimerasi III oloenzima, avviene il fenomeno dello switch delle polimerasi.
Il completamento della replicazione del DNA
Il termine della replicazione del DNA si ha quando il complesso di replicazione giunge alla fine del filamento stampo. I procarioti sono dotati di cromosomi che alla fine della sintesi di nuovo DNA, restano concatenati in una struttura definita “catenano”. È per questo motivo che dopo che il replisoma si stacca dall’acido nucleico, agiscono su di esso delle topoisomerasi II con il compito di eliminare i super avvolgimenti post-replicazione.
Il caso degli eucarioti risulta essere più complesso perché il filamento tardivo, a livello del telomero, presenterebbe un’estremità 3’ a ssDNA. Per risolvere questa problematica la cellula ha due possibilità:
Utilizzo di proteine primer: è appunto un primer proteico che fornendo un gruppo chimico -OH favorisce la corretta terminazione della replicazione.
Utilizzo della telomerasi: è un particolare tipo di DNA-polimerasi presentante un dominio RNA telomerasico grazie al quale non necessita di un primer e perciò può sintetizzare in modo autonomo, una sequenza che sia complementare al 3’ ssDNA.
Dal momento che la telomerasi potrebbe estendere di troppo i telomeri, questi si avvalgono di un meccanismo a feedback negativo grazie all’uso di specifiche proteine. Questa regolazione avviene in modo che in caso di estremità telomeriche già allungate, le proteine si stabiliscono sul filamento in gran numero impedendo per ingombro sterico l’associazione della telomerasi.
Fonti
“Biologia molecolare del gene”, Watson, Baker, Bell, Gann, Levine, Losick (a cura di), Zanichelli
“Biologia dei microrganismi”, Dehò, Galli (a cura di), Casa Editrice Ambrosiana
“I principi di biochimica di Lehninger”, Nelson, Cox (a cura di), Zanichelli
Immagine 1: https://elearning.uniroma1.it/pluginfile.php/653533/mod_resource/content/1/8%20lezione-replicazione%20%205%20aprile%202018%20%28Samanta%20Pino%29.pdf
Immagine 2: https://slideplayer.it/slide/10869897/
Immagine 3: https://www.wikiwand.com/en/DNA_clamp
Immagine 4: https://www.alamy.it/il-dna-elicasi-molecola-image65206635.html
Immagine 5: https://www.mdpi.com/2073-4425/4/1/1
Immagine 6: https://docplayer.it/123068659-La-replicazione-del-dna.html
Di: Gaia Bruno
Fonte Articolo: